科研进展
我所吴志强团队、杨剑团队合作在动物病毒组研究方面取得新进展
2025年11月20日,我所吴志强团队、杨剑团队联合在《Nucleic Acids Research》杂志在线发表了题为“ZOVER 2.0 : a virome-based platform for zoonotic and vector-borne viruses”的研究论文。
重要自然宿主与传播媒介携带的病毒组兼具高度复杂性与动态演化特性,持续对人类健康构成威胁。然而,传统病毒数据库多基于已知病毒基因组序列构建,难以全面呈现自然生态系统中病毒谱的真实多样性,尤其易遗漏低丰度或未受关注的病毒。为突破这一瓶颈,研究团队将前期ZOVER 1.0数据库升级到ZOVER 2.0版本,该版本系统整合了全球72项独立研究的5883个高质量宏基因组测序(mNGS)数据集,覆盖过去12年间采集的362种野生动物及媒介生物(含143种蝙蝠、125种鼠、48种蚊和46种蜱),样本遍及全球34个国家。同时,ZOVER 2.0创新采用“免组装”(assembly-free)分析策略,依托原始测序读段开展病毒分类与丰度估算,大幅提升低丰度病毒的检出灵敏度。经标准化数据处理,该平台共鉴定出110个病毒科、409个属及约4400个病毒种。与ZOVER 1.0的病毒分类对比分析显示,ZOVER 2.0从上述宏基因组数据中识别出30个新的哺乳动物相关病毒属。这些发现不仅拓展了野生动物病毒多样性的认知边界,更为新发传染病防控提供了关键数据支撑。
在此基础上,为打通病毒组数据挖掘到实用分析工具的转化链路,团队进一步开发了跨组学数据分析与可视化平台(http://www.mgc.ac.cn/ZOVER)。用户可通过该平台直观浏览、比较并解析不同宏基因组数据的病毒组差异,深度挖掘不同宿主类群、地理分布下的病毒组成特征与演化规律,还能与原平台的基因组数据开展联动分析,为未来依托多源异构数据开展新发传染病预警监测及检测技术研发等提供技术支撑。
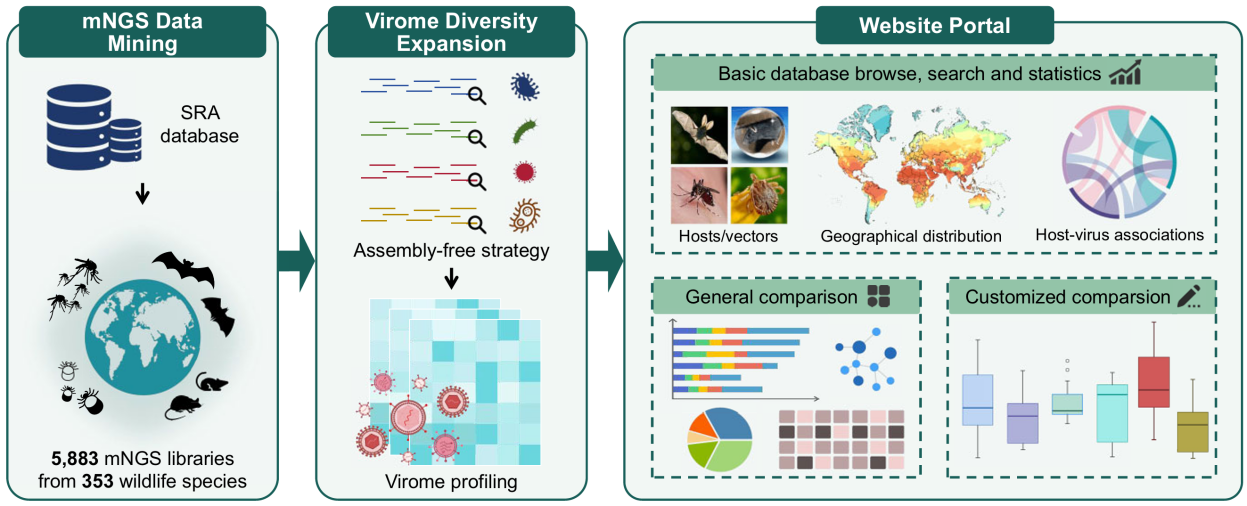
图. ZOVER 2.0疫源生物病毒谱研究示意图
我所刘博副研究员、博士研究生朱琳、周思宇助理研究员为论文的共同第一作者,吴志强研究员、杨剑研究员和陈立宏研究员为该论文的共同通讯作者。
